公開日: 04/21/2025
「マーカーの統計量」プラットフォームの別例
次のような手順で、遺伝子データから連鎖(linkage)に関する統計量を計算してみましょう。
1. [ヘルプ]>[サンプルデータフォルダ]>「Life Sciences」を選択し、「Genotypes Pedigree.jmp」と「Genotypes Pedigree Anno.jmp」を開きます。
2. [分析]>[遺伝学]>[マーカーの統計量]を選択します。
3. 「Markers」の開閉アイコンをクリックしてマーカーの列を表示します。
4. 最初の10個のマーカー列を選択し、[マーカー]をクリックします。
5. 同じ10個のマーカーを選択し、[with マーカー]をクリックします。
6. [注釈のデータテーブルを使用]チェックボックスをオンにします。
7. [OK]をクリックします。
8. 「注釈のデータテーブルを選択」ウィンドウで、「Genotypes Pedigree Anno.jmp」データテーブルを選択します。
9. 「注釈の指定」ウィンドウで、「マーカー」列を選択し、[マーカー変数]をクリックします。
10. 「遺伝子」列を選択し、[注釈 グループ]をクリックします。
11. 「物理位置」列を選択し、[注釈 位置]をクリックします。
12. [OK]をクリックします。
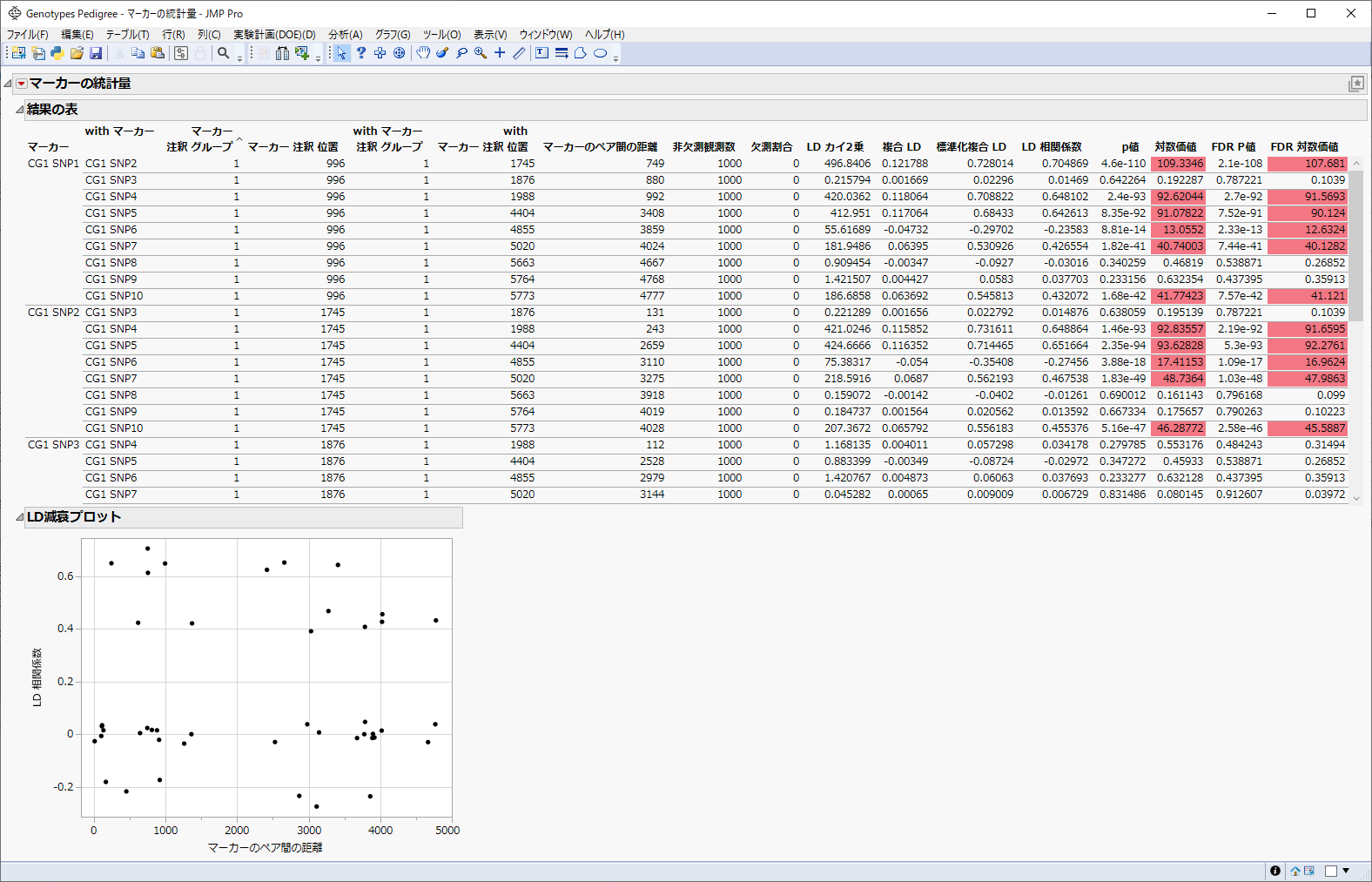
図3.4 連鎖不平衡の分析
「結果の表」に表示される各マーカーペアの値は、連鎖不平衡の強度を示しています。「LD減衰プロット」では、一般的には、連鎖不平衡は、マーカー間の距離が長くなるにつれて指数関数的に弱まっていきます。
より詳細な情報が必要な場合や、質問があるときは、JMPユーザーコミュニティで答えを見つけましょう (community.jmp.com).