1つの応答変数に対する変数重要度の例
この例では、予測プロファイルの[変数重要度の評価]オプションを使用して、ニューラルネットワークでモデル化された応答を予測する際に、どの変数が重要であるかを評価します。このオプションは、ニューラルネットワークのように、従来の仮説検定を行うのが難しいモデルに役立ちます。
なお、ニューラルネットワークのあてはめ、および、変数重要度の計算では、乱数を用いています。そのため、実際の結果は、以下と違ったものになります(おおむね同じになるはずです)。この例では、乱数を用いている処理が2個所あります。第1に、ニューラルネットワークをあてはめる際に、k分割交差検証を用います。この時、学習データと検証データに無作為に元のデータが分割されます。第2に、変数重要度の計算で、無作為に抽出した標本を使います。
1. [ヘルプ]>[サンプルデータフォルダ]を選択し、「Candy Bars.jmp」を開きます。
2. [分析]>[予測モデル]>[ニューラル]を選択します。
3. 「列の選択」リストで「カロリー」を選択し、[Y, 目的変数]をクリックします。
4. 「列の選択」リストで他のすべての連続尺度の列を選択し、[X, 説明変数]をクリックします。
5. [OK]をクリックします。
6. 「ニューラル」の「モデルの設定」パネルで、「検証法」の下のリストから[K分割]を選択します。
[K分割]を選択すると、「分割数」が表示され、デフォルト値の5に設定されます。
7. (オプション)「乱数シード値」に「123」と入力します。
メモ: ニューラルネットワークモデルでは、検証セットが無作為に抽出されるため、結果がいつも同じではありません。上記のものと同じ乱数シード値を入力すると、以下の結果を再現できます。
8. [実行]をクリックします。
9. 「モデル NTanH(3)」の赤い三角ボタンをクリックし、[プロファイル]を選択します。
レポートの末尾に「予測プロファイル」が表示されます。後で比較できるよう、因子の並び順を覚えておいてください。
因子間に相関関係があるため、その点を考慮して、重要度計算時の標本抽出法として[従属する標本再抽出の入力]を選択します。
10. 「予測プロファイル」の赤い三角ボタンをクリックし、[変数重要度の評価]>[従属する標本再抽出の入力]を選択します。
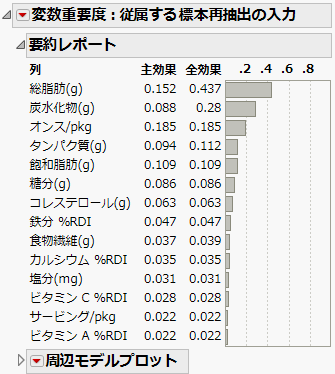
図3.27 「従属する標本再抽出の入力」レポート
「変数重要度:従属する標本再抽出の入力」レポートが表示されます。「予測プロファイル」のセルの並び順が、レポートの「全効果」の値の大きい順に変化している点を確認してください。Figure 3.27では、「全効果」の値から、「総脂肪(g)」と「炭水化物(g)」が応答の予測値に大きい影響力を持つ因子だと判断できます(それぞれ0.437と0.28)。同様に、「オンス/pkg」と「タンパク質(g)」は、それらより少し小さい値ではあるものの、影響力のある因子です(それぞれ0.185と0.112)。
因子間に相関があると仮定して求めた重要度と、因子が相互に独立であると仮定して求めた重要度を比較してみてもよいでしょう。
11. 「予測プロファイル」の赤い三角ボタンをクリックし、[変数重要度の評価]>[独立な標本再抽出の入力]を選択します。
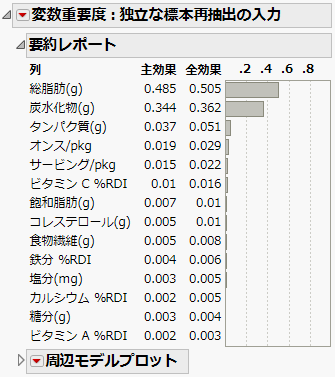
図3.28 「独立な標本再抽出の入力」レポート
因子間の相関がなく、分布が一様分布でない場合には、この[独立な標本再抽出の入力]オプションを用いるのが良いでしょう。「変数重要度:独立な標本再抽出の入力」レポートは、「総脂肪(g)」と「炭水化物(g)」は、依然として予測値に大きい影響力を持つ(それぞれ0.505と0.362)ことを示しています。同様に、「タンパク質(g)」と「オンス/pkg」は、それらに比べるとかなり小さい値ではあるものの、効果のある因子です(それぞれ0.05と0.03)。Figure 3.27と比較すると、「総脂肪(g)」と「炭水化物(g)」の「全効果」の重要度はわずかに増加していますが、「オンス/pkg」と「タンパク質(g)」は大幅に減少しています。これより、標本再抽出手法によって変数重要度の値にばらつきがあることがわかります。