「マーカーの統計量」プラットフォームの起動
手持ちの実験データでマーカーの統計量を計算するには、[分析]>[遺伝学]>[マーカーの統計量]を選択して「マーカーの統計量」プラットフォームを起動します。
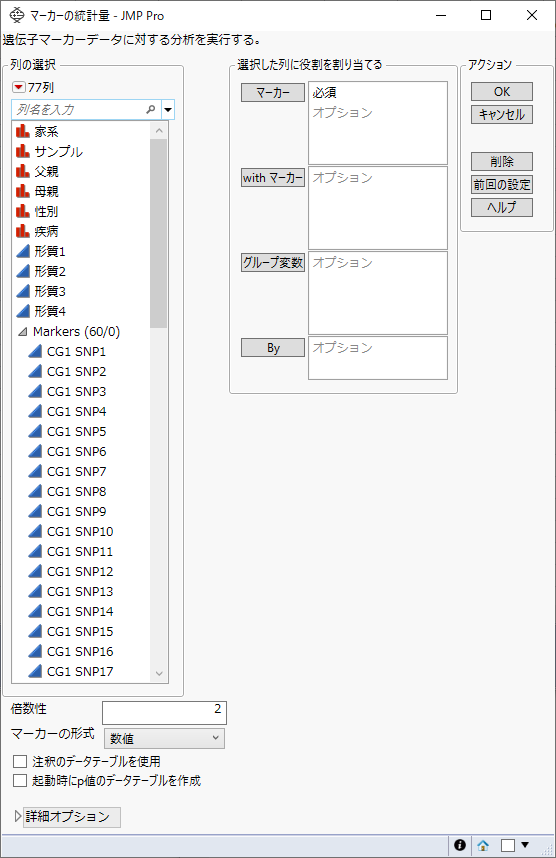
図3.3 「マーカーの統計量」起動ウィンドウ
「列の選択」の赤い三角ボタンのメニューにあるオプションについては、『JMPの使用法』の“Column Filter Menu”を参照してください。
マーカー
分析対象のマーカーを指定するには、該当するマーカー列を選択し、[マーカー]をクリックします。
with マーカー
[マーカー]で指定したマーカーと他のマーカーをペアにして連鎖不平衡を調べるには、該当するマーカー列を選択して[with マーカー]をクリックします。
グループ変数
指定の列の各水準に割り当てられた行を個別に分析します。結果は1つの表・レポートにまとめて表示されます。
By
By変数を指定すると、その変数の水準ごとに個別のレポートが作成されます。複数のBy変数を割り当てた場合は、それらのBy変数の水準の組み合わせごとに個別にレポートが作成されます。
倍数性
調査対象である生物の倍数性(ploidy)を指定できます。
マーカーの形式
入力データにおいて、マーカーがどのような形式で保存されているかを指定します。
この「マーカーの形式」オプションはJMP18で追加された機能です。このオプションにより、数値以外で保存されているマーカーデータも使用できるようになりました。数値ではないマーカーデータは、プラットフォームを実行した時点で再コード化され、数値に再コード化されたデータが元データに追加されます。数値マーカーに変換されたデータは、数値形式のマーカーデータを必要とする他の「遺伝学」プラットフォームや、数値列を分析対象とするJMPの他のプラットフォームで使用できます。
• マーカーが数値形式でコード化されている場合は、[数値]を選択します。通常、この形式では、2倍体の生物において、頻度の少ない方の(つまりマイナーアレルの方の)ホモ接合を「2」、ヘテロ接合を「1」で表します。そして、頻度の多い方のアレルのホモ接合は「0」で表します。多倍体サンプルの場合、列の遺伝子型は0, 1, 2, …, pという値をとります(ここで、pは倍数)。
• 二倍体サンプルのマーカーがA, B, H(ここでA = AA, B = BB, H = AB)としてコード化されている場合は、[一文字]を選択します。
• p倍数のサンプルがApBpとしてコード化されている場合は、[文字]を選択します(p = 倍数。例: 4倍体サンプルの場合はAAAA, AAAB, AABB, ABBB, BBBB)。
• 二倍体サンプルがIUPACコード(A, C, G, T, R, Y, S, W, K, M, +, 0, -, N)でコード化されている場合は、[1コードのヌクレオチド]を選択します。
• 二倍体サンプルが2文字(AA, CC, GG, TT, AC, AGなど)でコード化されている場合は、[ヌクレオチド]を選択します。
注釈のデータテーブルを使用
別のデータテーブルに含まれている注釈(アノテーション)を参照できます。[OK]をクリックすると、注釈(アノテーション)のデータテーブルの名前と場所を指定するためのウィンドウが開きます。
起動時にp値のデータテーブルを作成
関連するマーカー統計量について、p値のデータテーブルを作成します。このデータテーブルは、「マーカーの統計量」レポートの「結果の表」にリンクされます。
スレッドを使用しない
このオプションをオンにすると、マルチスレッドを使わないようになります。計算速度を速くしたい場合は、このオプションをオフにしてください。
「マーカーの統計量」プラットフォームに必要なデータ形式
JMPで処理や統計分析を行うほとんどの場合において、分析対象のデータテーブルが特定のデータ構造で作成されていることを前提としています。JMPでは、縦長形式(tall; トール)と横長形式(wide; ワイド)のデータ構造が区別されます。縦長形式のデータテーブルは、サンプルが列で、分子に関する情報(マーカー・遺伝子・クローン・タンパク質・代謝物など)が行です。一方、横長形式のデータテーブルは、縦長のデータテーブルを転置したもので、サンプルが行、分子に関する情報が列となります。
分析する入力データセットを指定する際には、ソフトウェアに必要な形式を知っておくことが重要です。「マーカーの統計量」プラットフォームでは、分析対象のデータテーブルを横長形式にしておく必要があります。[テーブル]メニューの[転置]オプションを使用すると、縦長形式のデータテーブルを横長形式に、または横長形式のデータテーブルを縦長形式に変換できます。