발행일 : 03/10/2025
 다변량 임베딩의 추가 예
다변량 임베딩의 추가 예
이 예에서는 다변량 임베딩의 예와 동일한 데이터 집합을 사용하지만 데이터를 2차원이 아니라 3차원으로 축소합니다. 또한 UMAP 방법과 t-SNE 방법 간의 결과를 비교합니다.
1. 도움말 > 샘플 데이터 폴더를 선택하고 Cytometry.jmp를 엽니다.
2. 분석 > 다변량 방법 > 다변량 임베딩을 선택합니다.
3. CD3 ~ MCB를 선택하고 Y, 열을 클릭합니다.
4. "방법"에 "t-SNE"를 지정합니다.
5. "출력 차원" 옆에 "3"을 입력합니다.
6. (선택 사항) "난수 시드값" 옆에 "1234"를 입력합니다.
참고: 위의 시드값을 입력하면 이 예에 표시된 결과를 재현할 수 있습니다.
7. 대화상자 열린 채 유지를 선택합니다.
8. 확인을 클릭합니다.
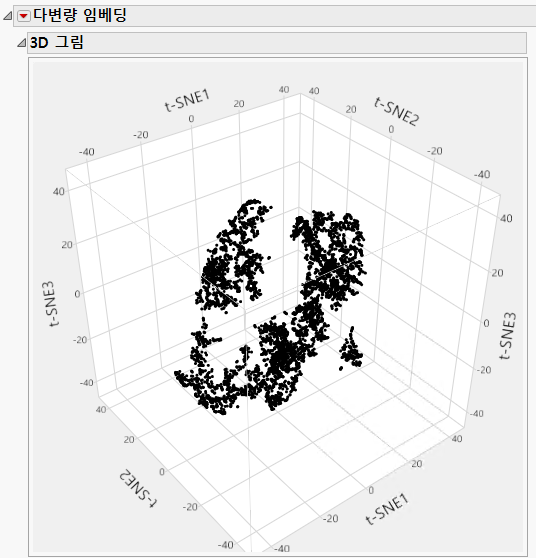
그림 11.4 3D t-SNE 그림의 예
9. 시작 창에서 "방법"을 "UMAP"으로 변경합니다.
10. 확인을 클릭합니다.
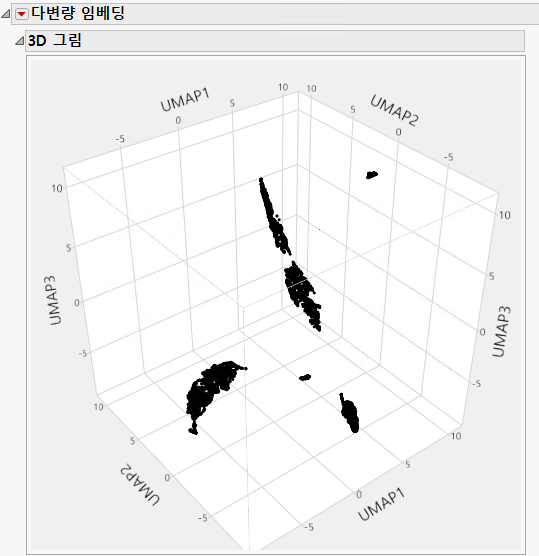
그림 11.5 3D UMAP 그림의 예
각 3D 그림에는 해당하는 차원 축소 방법에서 처음 세 개의 임베딩 성분에 대한 좌표가 표시됩니다. UMAP 방법과 t-SNE 방법을 비교하면 UMAP 방법을 사용했을 때 데이터가 더 조밀하게 군집화되면서도 계산 시간이 적게 걸렸습니다. Figure 11.4의 3D t-SNE 그림과 비교할 때 3D UMAP 그림에서 더 뚜렷한 군집을 볼 수 있습니다.
팁: 격자를 회전하여 군집을 더 명확하게 볼 수 있습니다.
더 많은 정보를 원하십니까? 질문이 있습니까? JMP 사용자 커뮤니티에서 답변 받기 (community.jmp.com).