启动“标记亲缘”平台
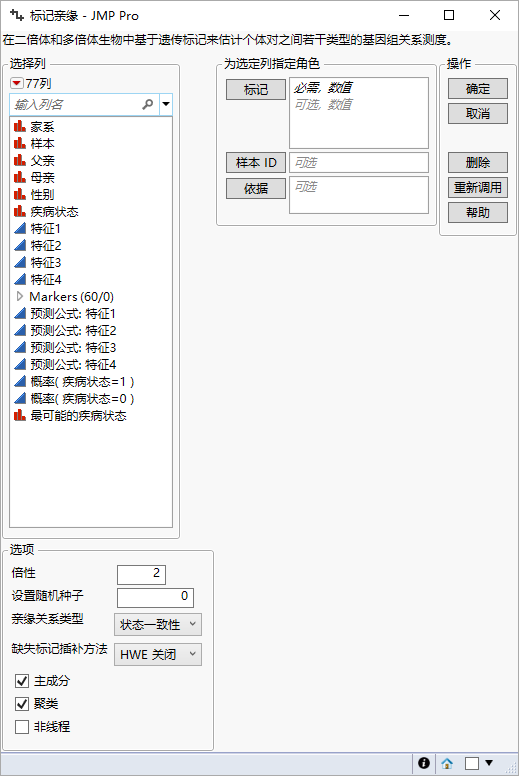
要估计数据表中个体对之间的遗传关系的方矩阵,通过选择分析 > 遗传学 > 标记亲缘启动“标记亲缘”平台。
图 5.3 “标记亲缘”启动窗口
标记
选择所需的标记列,然后点击标记指定要分析的标记。
样本 ID
使用该选项指定一个变量,该变量的值可以提供每行的唯一标识符。
注意:仅允许指定一个变量。
依据
为“依据”变量的每个水平生成单独报表。若指定了多个“依据”变量,将为“依据”变量水平的每种可能组合生成单独的报表。
倍性
允许您指定所研究的实验生物体的倍性水平。注意:该值必须是偶数
设置随机种子
使用该选项可指定一个非负整数以启动随机数流。不同的值会生成不同的算法结果。
亲缘关系类型
使用该选项指定用于评估样本(行)之间关系的测量值的类型。
• 选择状态一致性检查标记标识对个体亲缘的影响。
• 选择加性检查加性标记对个体亲缘的影响。
• 选择显性检查标记显性对个体亲缘的影响。
• 选择上位性检查标记交互作用对个体亲缘的影响。
加性类型
使用该选项指定一种方法来评估加性标记对个体亲缘的影响。
• 选择二倍体方法 1 使用 Van Rayden 第一方法 (Van Raden, et al.,2008) 来计算关系矩阵。(P.M. Van Raden, “Efficient Methods to Compute Genomic Predictions”.J.Dairy Sci.91:4414-4423, 2008)
• 选择二倍体方法 2 使用 Van Raden, et al., 2008 第二方法。(P.M. VanRaden, “Efficient Methods to Compute Genomic Predictions”.J.Dairy Sci.91:4414-4423, 2008)
• 选择多倍体 使用 Batista et al.(2022) 方法。该方法使用倍性和等位基因剂量的估计值构建加性效应的协方差矩阵。Batista, L.G., Mello, V.H., Souza, A.P. et al. Genomic prediction with allele dosage information in highly polyploid species.Theor Appl Genet 135, 723–739 (2022). https://doi.org/10.1007/s00122-021-03994-w
注意:仅当将加性选作亲缘关系类型,或选择了上位性并且将加性 - 加性或显性 - 加性选作上位性类型时,该选项才可用。
显性类型
使用该选项指定一种方法来评估标记显性对个体亲缘的影响。
• 选择二倍体方法 1 使用 Su, et al.(2012) 方法。(Su G, Christensen OF, Ostersen T, Henryon M, Lund MS.Estimating additive and non-additive genetic variances and predicting genetic merits using genome-wide dense single nucleotide polymorphism markers.PLoS One.2012;7(9):e45293. doi: 10.1371/journal.pone.0045293.Epub 2012 Sep 13.PMID: 23028912; PMCID: PMC3441703.)
• 选择二倍体方法 2 使用 Vitezica, et al.(2012) 方法。该方法使用个体间显性基因组关系的矩阵。该矩阵在混合模型上下文中可用于估计总体中的显性方差。Zulma G Vitezica, Luis Varona, Andres Legarra.2013.On the Additive and Dominant Variance and Covariance of Individuals Within the Genomic Selection Scope, Genetics 195: 1223–1230. (https://doi.org/10.1534/genetics.113.155176)
• 选择多倍体使用 Batista et al.(2022) 方法。该方法使用倍性和等位基因剂量的估计值构建显性效应的协方差矩阵。Batista, L.G., Mello, V.H., Souza, A.P. et al. Genomic prediction with allele dosage information in highly polyploid species.Theor Appl Genet 135, 723–739 (2022). https://doi.org/10.1007/s00122-021-03994-w
注意:仅当将显性选作亲缘关系类型,或选择了上位性并且将显性 - 加性或显性 - 显性选作上位性类型时,该选项才可用。
上位性类型
使用该选项指定一种方法来评估标记交互作用对个体亲缘的影响。
• 选择加性 - 加性计算对话框中指定的加性类型与其自身之间的交互作用。例如,当“加性类型”为“方法 1”时,它会计算该关系类型与其自身之间的交互作用。
• 选择显性 - 加性计算对话框中指定的加性类型与显性类型之间的交互作用。例如,当“加性类型”为“方法 1”且“显性类型”为“方法 1”时,它会计算这两种关系类型之间的交互作用。
• 选择显性 - 显性计算对话框中指定的显性类型与其自身之间的交互作用。例如,当“显性类型”为“方法 1”时,它会计算该关系类型与其自身之间的交互作用。
注意:仅当将上位性选作亲缘关系类型时,该选项才可用。
缺失标记插补方法
当您的数据缺少标记数据时,该平台不会运行。因此,必须对任何缺失数据进行插补。使用该选项可指定如何插补缺失值。
‒ 选择 HWE 关闭可使用多项式分布中的随机抽取来插补缺失基因型,其中每个基因型类的频数都设置为数据中的观测频数。
‒ 选择 HWE 打开可使用多项式分布中的随机抽取来插补缺失基因型,其中每个基因型类的频数都设置为 Hardy-Weinberg 平衡假设下的期望频数。
‒ 选择随机随机指定一个可接受值(0, 1, 2, ..., K(其中 K 是倍性水平))。
‒ 选择指定可使用介于 0 和倍性数之间的指定整数来插补缺失基因型。
插补值
当您在缺失标记插补方法中选择“指定”选项时,在文本框中指定 0 与倍性数之间的值。
此处列出的值假定为二倍体生物体;对倍性较高的生物体相应地调整这些值。
主成分
选中该框可计算并绘制方矩阵上的主成分。
聚类
选中该框可按照标记亲缘对个体进行聚类。
非线程
禁止多线程。取消选择该选项可提高计算速度。
“标记亲缘”平台所需的数据格式
JMP 中的大多数过程认定输入表具有特定的数据结构。JMP 区分高型和宽型数据集。高型数据表以样本作为列,分子实体(例如,标记、基因、克隆、蛋白质或代谢物)作为行,而宽型数据表是高型数据表的转置,以样本作为行,分子实体作为列。
为过程指定输入数据集时,了解所需的格式十分重要。“标记亲缘”需要宽型数据表。“表”菜单下的“转置”平台使您可以在高型和宽型格式之间转换您的数据。
标记数据必须采用单列数值格式编码。通常,在这种格式中,最不常见或次要等位基因的两倍体个体纯合在表中用 2 表示,而杂合子用 1 表示。最常见的等位基因的纯合子用 0 表示。