「マーカーの関係性」の例
次のような手順で、手持ちの遺伝子データをもとに交配をシミュレートしてみましょう。
1. [ヘルプ]>[サンプルデータフォルダ]>「Life Sciences」を選択し、「Genotypes Pedigree.jmp」と「Genotypes Pedigree Anno.jmp」を開きます。
2. 「Genotypes Pedigree.jmp」データテーブルをクリックします。
3. [分析]>[遺伝学]>[マーカーの関係性]を選択します。
4. 「Markers (60/0)」を選択し、[マーカー]をクリックします。
5. 「性別」を選択し、[By]をクリックします。
6. 「倍数性」に「2」と入力します。
7. 「乱数シード値」に「12345」と入力します。
8. 「ゲノム関係性の種類」を[相加性]に設定します。
9. 「相加的関係の種類」に[二倍体法1]を指定します。
10. 「欠測マーカーの補完法」を[ランダム]に設定します。
11. 「主成分分析」と「クラスター分析」の両方のボックスをチェックしたままにします。
12. [OK]をクリックします。
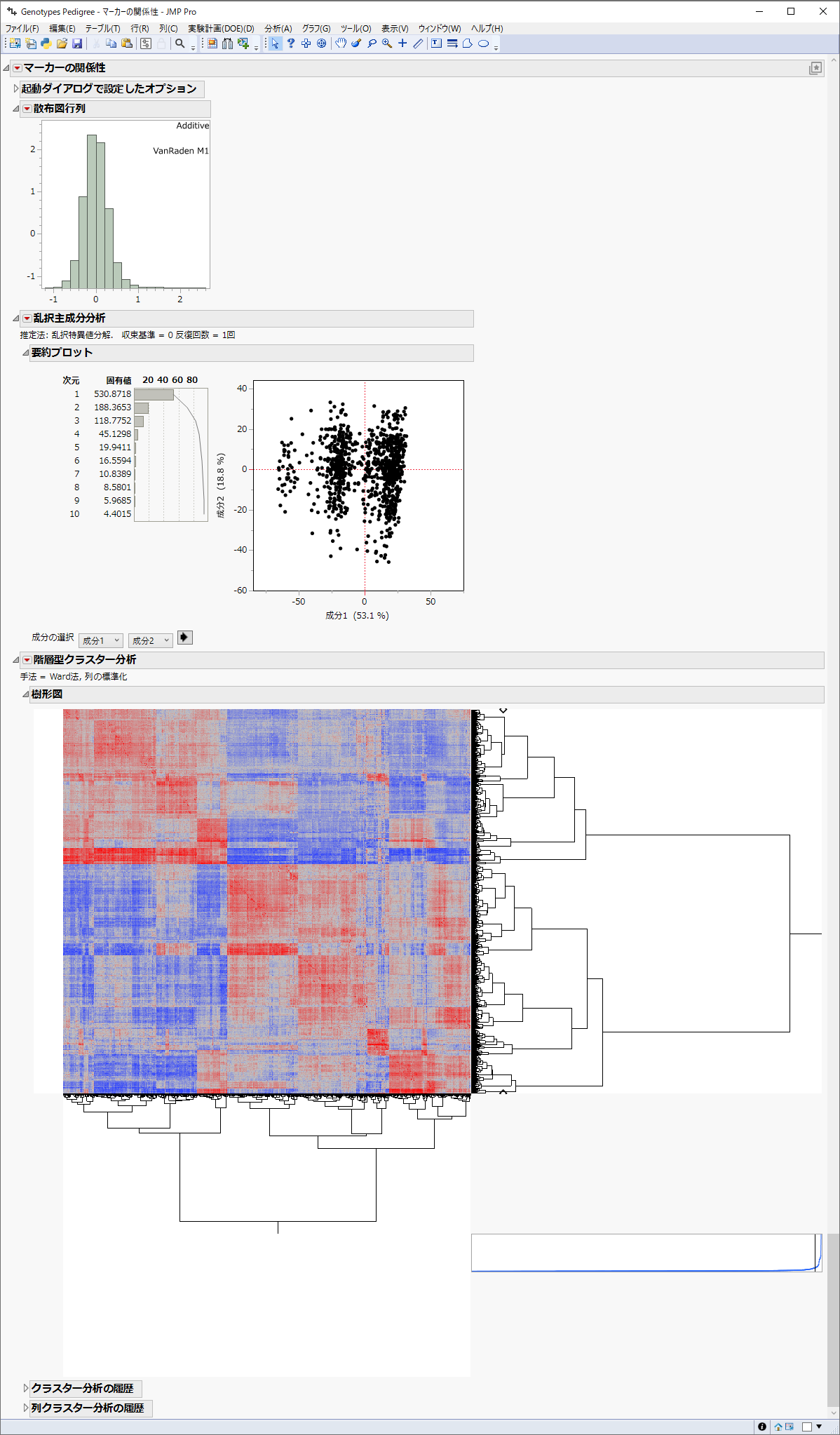
「マーカーの関係性」レポートが図5.1のように生成されます。
図5.2 マーカーの関係性
「マーカーの関係性」レポートは、各性別(MおよびF)について1回ずつ実行され、以下の情報が含まれます。結果は男女別に表示されます。
• 「起動ダイアログで設定したオプション」は、シミュレーションの設定と処理時間をまとめたものです。
• 「散布図行列」には、ヒストグラムが表示されます(エピスタシスを推定した場合は散布図行列が表示されます)。ヒストグラムが右に偏っている(つまり、値が大きい)場合、ほとんどの個体が遺伝的に強い類似性を持っていることを示します。逆に、ヒストグラムが左に偏っている(つまり、値が小さい)場合、ほとんどの個体が遺伝的に弱い類似性を持っていることを示します。
• 主成分分析プロットは、関係性によってグループ化した個体のクラスターを見つけ出すのに役立ちます。ここに示した例では、個体は明確なクラスターを形成していないように見えます。
• クラスター分析プロットは、関係性によってグループ化するもう1つの方法です。赤い三角形で、[変数間クラスター]オプションをクリックすると、ヒートマップが作成されます。このヒートマップは、すべての個体間の関係を視覚的に調べるのに役立ちます。