로버스트 적합의 예
반응 변수 선별 플랫폼의 "로버스트" 옵션을 사용하여 이상치가 많은 반응을 분석합니다. 이상치는 기존 검정의 오차 분산을 부풀려 유의한 효과를 알아보기 어려울 수 있으므로 로버스트 적합을 사용해야 합니다.
1. 도움말 > 샘플 데이터 폴더를 선택하고 Drosophila Aging.jmp를 엽니다.
2. 분석 > 선별 > 반응 변수 선별을 선택합니다.
3. 연속형 열을 모두 선택하고 Y, 반응을 클릭합니다.
4. Line을 선택하고 X를 클릭합니다.
5. "고급 옵션" 옆의 회색 삼각형을 클릭합니다.
6. 로버스트를 선택합니다.
7. 확인을 클릭합니다.
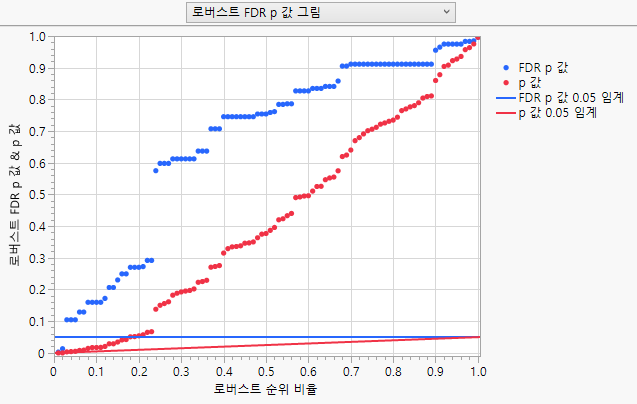
그림 24.12 Drosophila 데이터에 대한 로버스트 FDR p 값 그림
0.05 미만의 빨간색 점으로 표시된 것처럼 조정되지 않은 로버스트 p 값을 사용하여 많은 검정이 유의합니다. 그러나 로버스트 FDR p 값에 따르면 두 가지 검정만 유의합니다. 이 두 점은 FDR LogWorth 값을 보여 주는 그림에서 더 쉽게 식별됩니다.
8. "반응 변수 선별"의 그림 제목 목록을 클릭하고 로버스트 FDR LogWorth 대 효과 크기를 선택합니다.
9. 로버스트 FDR LogWorth 값이 2를 초과하는 점을 클릭하고 마우스 오른쪽 버튼을 클릭한 후 행 라벨을 선택합니다.
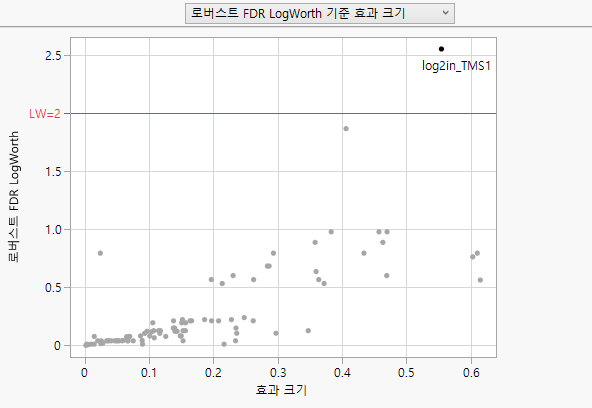
그림 24.13 Drosophila 데이터에 대한 로버스트 LogWorth 대 효과 크기
빨간색 선(2) 위의 점은 유의 수준이 0.01(-log10(0.01) = 2) 미만입니다. 가로선(약 1.3)은 0.05 유의 수준에 해당합니다.
10. "반응 변수 선별"의 그림 제목 목록을 클릭하고 로버스트 FDR LogWorth 대 FDR LogWorth를 선택합니다.
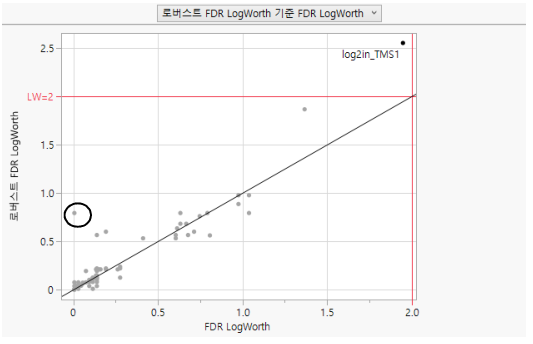
그림 24.14 Drosophila 데이터에 대한 로버스트 FDR LogWorth 대 FDR LogWorth
반응에 대한 로버스트 검정이 일반 검정과 동일할 경우 해당 점은 Figure 24.14에서와 같이 대각선 위에 분포합니다. 그림에서 원 안의 점은 로버스트 LogWorth 값이 LogWorth 값을 초과하므로 선에서 떨어져 있습니다.
11. 그림에서 이 점을 클릭하여 선택합니다.
12. "결과 테이블"에서 이 점에 해당하는 행을 찾습니다.
반응 log2in_CG8237의 p 값은 0.9568이고 로버스트 p 값은 0.0176입니다.
13. "반응 변수 선별"의 빨간색 삼각형을 클릭하고 선택한 항목 적합을 선택합니다.
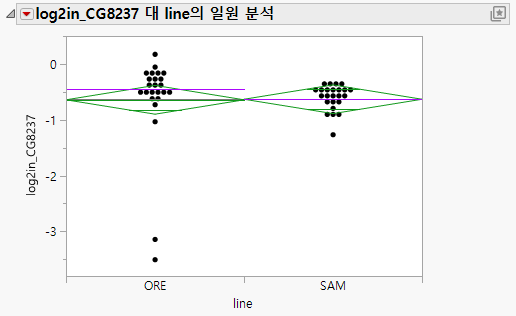
반응 log2in_CG8237에 대한 일원 분석이 포함된 "선택한 항목 적합" 보고서가 표시됩니다. 그림에 ORE line에 대한 두 개의 이상치가 표시됩니다(Figure 24.15). 이러한 이상치는 로버스트 검정과 일반 검정의 결과가 서로 다른 이유를 나타냅니다. 이상치는 비로버스트 검정의 오차 분산을 부풀리므로 유의한 효과를 알아보기가 더 어렵습니다. 반면, 로버스트 적합은 이러한 이상치의 비중을 낮추어 오차 분산에 대한 기여도를 줄입니다.
그림 24.15 log2in_CG8237에 대한 일원 분석