重复测量示例
重复测量示例
考虑 Cholesterol Stacked.jmp 样本数据表。执行了一项研究来检验两种新胆固醇药物相对于对照药物的效果。将具有高胆固醇的二十名患者随机分配给四个治疗方案之一(两个实验药物、对照药物和安慰剂)。在研究期间分别在以下六个时间测量每名患者的总胆固醇:四月、五月和六月的第一天的早上和下午。您想了解这两种新药物对于降低胆固醇是否有效以及时间和治疗是否有交互作用。
 背景
背景
过去一直使用以下两种方法来分析这类设计:
• 多元方差分析 (MANOVA)
• 进行 Huynh-Feldt (1976) 或 Greenhouse-Geisser (1959) 校正的时间裂区一元方差分析 (ANOVA)
在“拟合模型”中使用“多元方差分析”特质可提供这两个选项。这两个选项是对协方差结构建模的两个极端。MANOVA 分析假定一个非结构化协方差结构,其中单独估计所有方差和协方差。独立的时间裂区分析假定所有误差是独立的。在高斯数据案例中,这等价于假定复合对称性协方差结构。
这两个模型可能得到有关治疗效应的截然不同的结论。当您假定复杂的协方差结构时,使用数据中的信息来估计协方差参数。若您拟合过多的协方差参数,可能会过拟合模型。对重复测量数据建模时,必须找到平衡处理这些问题的协方差结构。
• 模型过拟合时,检验差值的功效小于您假定相对简单的协方差结构时的功效。
• 模型拟合不足时,将失去对第一类错误的控制。在一些情况下,这导致拒绝率增大。在另一些情况下,拒绝率可能由于方差增大而减小。
 协方差结构
协方差结构
“混合模型”特质拟合各种协方差结构。对于时间上的重复测量,Toeplitz 协方差结构和一阶自回归 (AR(1)) 协方差结构通常提供合适的相关性结构。这些结构允许有相关性的观测而不会过拟合模型。AR(1) 假定一个公共方差参数,而具有不等方差的 Toeplitz 协方差矩阵则为每个重复测量变量单元估计唯一的方差。请参见重复协方差结构要求。
在该示例中,您拟合四个协方差结构。观测次数 J 为 6。
• 协方差结构:非结构化。非结构化模型一共拟合的协方差参数数目为 J(J+1)/2。在该示例中,模型拟合 21 个方差。
• 协方差结构:残差。“残差”模型等价于通常的方差分量结构。在该示例中,模型拟合两个方差。
• 协方差结构:Toeplitz。Toeplitz 模型拟合 2J-1 个协方差参数。在该示例中,模型拟合 11 个方差。
• 协方差结构:AR(1)。该模型拟合两个协方差参数。一个参数确定方差,另一个参数确定协方差如何随时间变化。
您使用 AICc 评估模型拟合效果。还可以使用 BIC 准则。在该示例中,这两个准则选择同一模型。您选择一个最佳协方差结构,然后继续执行其他分析:
提示:完成该示例中的步骤时,请保持“拟合模型”启动窗口打开。
 数据结构
数据结构
Cholesterol.jmp 数据表采用通常用于记录重复测量数据的格式。要使用“混合模型”特质分析这些数据,每个胆固醇测量值需要在自己的行中,如 Cholesterol Stacked.jmp 中所示。要构造 Cholesterol Stacked.jmp,请使用“表”>“堆叠”来堆叠 Cholesterol.jmp 中的数据。
使用公式构造的堆叠表中的天数列。天数列给出测量胆固醇时已进行研究的天数。建模类型为连续。这是必要的,因为“AR(1)”协方差结构要求重复效应是连续的。
 协方差结构:非结构化
协方差结构:非结构化
首先使用“非结构化”协方差结构来拟合模型。
1. 选择帮助 > 样本数据库,然后打开 Cholesterol Stacked.jmp。
2. 选择分析 > 拟合模型。
3. 选择保持对话框打开,以便您可以在下一示例中返回到启动窗口。
4. 选择 Y 并点击 Y。
5. 从“特质”列表中选择混合模型。
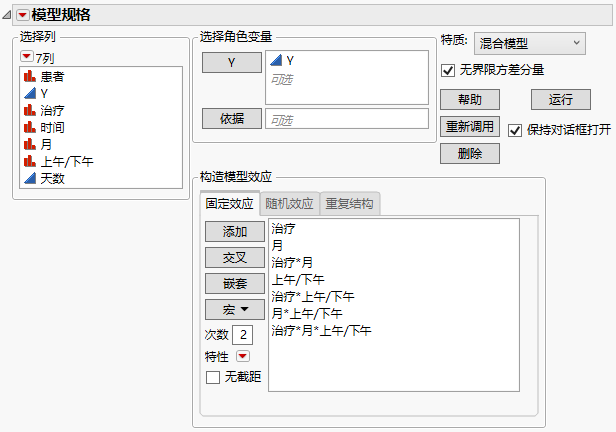
6. 选择治疗、月和上午/下午,然后选择宏 > 完全析因。
图 8.12 显示完成的“固定效应”选项卡的“拟合模型”启动窗口
7. 选择重复结构选项卡。
8. 从“结构”列表中选择非结构化。
9. 选择时间,然后点击重复。重复列定义对象内的重复测量值。
10. 选择患者并点击对象。
注意:“非结构化”协方差模型不允许重复结构变量采用重复值。在该示例中,假定对象在治疗内嵌套且在每个治疗内使用值 1、2、3、4 和 5 对患者进行了编号。运行该分析时将显示一个警告。您需要重新对患者编号以使重复变量的每个值具有不同的标识符。或者,您需要在数据表中创建一列表示治疗内嵌套并将该效应作为“对象”输入。
图 8.13 显示完成的“重复结构”选项卡的“拟合模型”启动窗口
11. 点击运行。
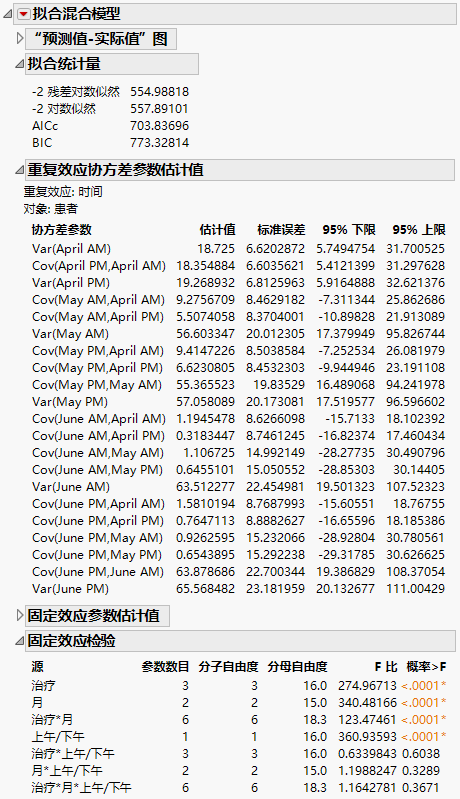
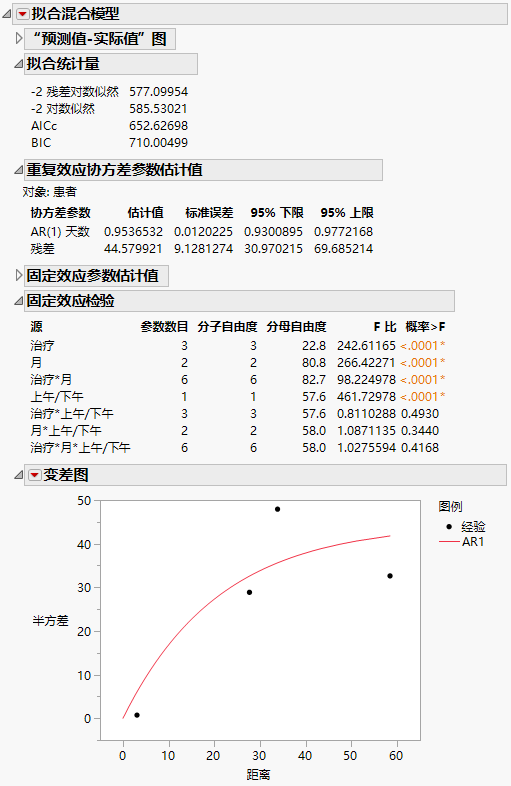
“拟合混合模型”报表显示在Figure 8.14 中。 因为要使用 AICc 或 BIC 来比较三个模型,您关注“拟合统计量”报表。非结构化模型的 AICc 为 703.84。
“重复效应协方差参数估计值”报表显示所有 21 个协方差参数的估计值。如您所希望的,在时间上挨得更近的观测值比时间相隔较远的观测值具有更高的协方差。此外,方差随时间增加。
图 8.14 “拟合混合模型”报表 -“非结构化”协方差结构
 协方差结构:残差
协方差结构:残差
拟合裂区模型时,“残差”协方差结构是合适的。
1. 完成协方差结构:非结构化中的step 1到step 7。
2. 在重复结构选项卡上,从“结构”列表中选择残差。
3. 若您接上例继续操作,请删除时间和患者。
否则将显示一条警告:“当选择残差协方差结构时忽略重复列和对象列。”系统提供您选项来点击确定以继续分析。
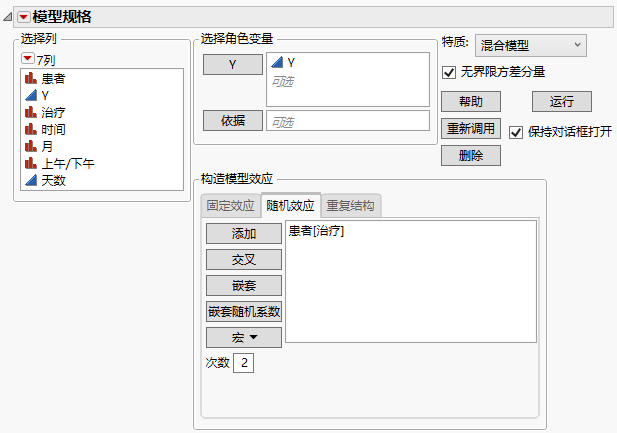
4. 选择随机效应选项卡。
5. 选择患者,然后点击添加。
6. 选择“随机效应”区域中的患者,选择治疗列,然后点击嵌套。
图 8.15 显示完成的“随机效应”选项卡的“拟合模型”启动窗口
7. 点击运行。
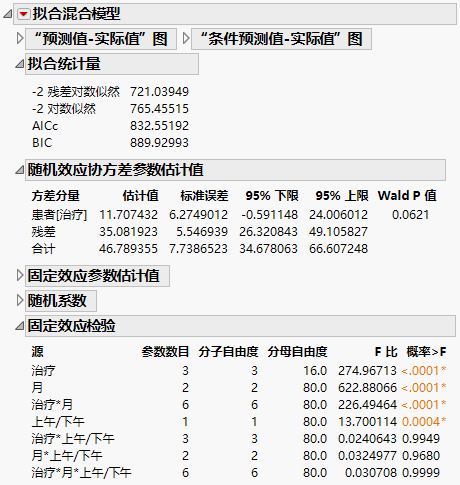
“拟合混合模型”报表显示在Figure 8.16 中。“拟合统计量”报表显示“残差”模型的 AICc 是 832.55,而“非结构化”模型的 AICc 为 703.84。
两个协方差参数的估计值显示在“随机效应协方差参数估计值”报表中。这些是“‘患者’嵌套在‘治疗’内”方差的估计值和“残差”方差的估计值。
图 8.16 “拟合混合模型”报表 - “剩余误差”协方差结构
 协方差结构:Toeplitz
协方差结构:Toeplitz
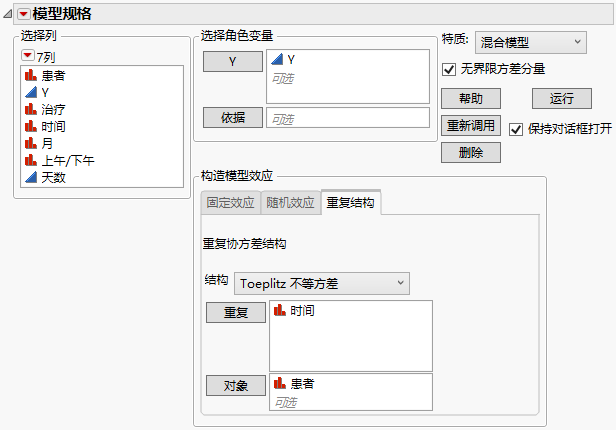
使用“Toeplitz 不等方差”结构拟合模型。
1. 完成协方差结构:非结构化中的step 1到step 6。
2. 若您接上例继续操作,请选择随机效应选项卡上的患者[治疗],然后点击删除。
若您同时包含随机效应和重复效应,估计这两个效应的数据通常不足。
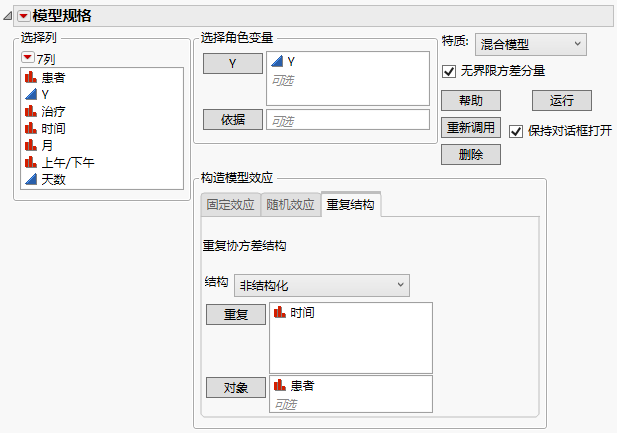
3. 选择重复结构选项卡。
4. 从“结构”列表中选择 Toeplitz 不等方差。
5. 选择时间,然后点击重复。
6. 选择患者并点击对象。
图 8.17 显示完成的“重复结构”选项卡的“拟合模型”启动窗口
7. 点击运行。
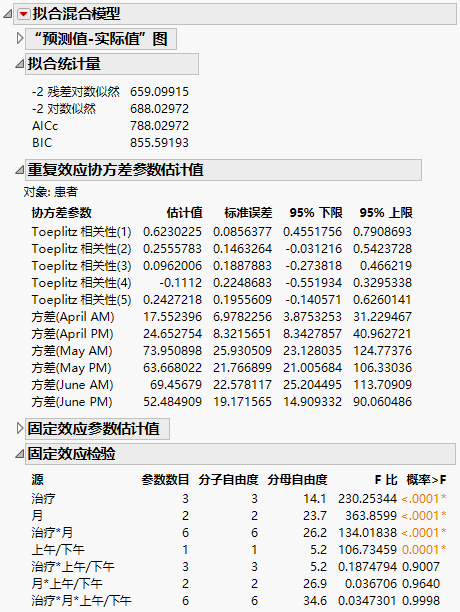
图 8.18 “拟合混合模型”报表 - Toeplitz 不等方差结构
注意:JMP 中的“混合模型”特质报告相关性,而 SAS 中的 PROC MIXED 报告协方差。
“拟合统计量”报表显示具有不等方差的 Toeplitz 模型的 AICc 为 788.03。将该数字与残差模型的 832.55 以及非结构化模型的 703.84 进行比较。
“Toeplitz 不等方差”结构需要估计 11 个协方差参数。这些估计值显示在“重复效应协方差参数估计值”报表中。显示 Toeplitz 相关性估计值,后跟每个时间点的方差估计值。有关如何参数化该矩阵的信息,请参见重复测量。
 协方差结构:AR(1)
协方差结构:AR(1)
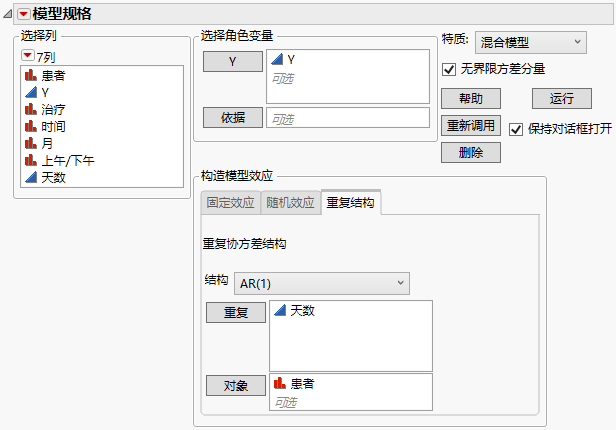
最后,拟合“AR(1)”结构。
1. 完成协方差结构:非结构化中的step 1到step 6。
2. 若是继续前一示例,请在重复框中选择时间,然后点击删除。
AR(1) 需要重复值的连续变量。
3. 从“结构”列表中选择 AR(1)。
4. 选择天数,然后点击重复。
图 8.19 显示完成的“重复结构”选项卡的“拟合模型”启动窗口
5. 点击运行。
“拟合混合模型”报表显示在Figure 8.20 中。“拟合统计量”报表显示 AR(1) 模型的 AICc 为 652.63。将该数字与残差模型的 832.55、非结构化模型的 703.84、Toeplitz 不等方差模型的 788.03 进行比较。根据 AICc 准则,AR(1) 模型在这四个模型中是最佳的。
“AR(1)”结构需要估计两个协方差参数。这些估计值显示在“重复效应协方差参数估计值”报表中。“AR(1)天数”参数估计值是“AR(1)”结构中相关性参数 ρ 的估计值。
“变差图”显示经验半方差和 AR(1) 模型的曲线。因为天数只有五个非零值,只可能有四个距离分类,因此只显示四个点。“AR(1)”结构看起来是合适的。要探索其他结构,请从“变差图”红色小三角菜单中选择相应选项。有关“变差图”选项的详细信息,请参见变差图。
图 8.20 “拟合混合模型”报表 - “AR(1)”协方差结构
 使用 AR(1) 结构进行进一步分析
使用 AR(1) 结构进行进一步分析
因为“AR(1)”模型给出最佳拟合,您采用它作为模型并继续分析。“固定效应检验”报表指示“治疗”和“月”之间有显著交互作用以及存在“上午/下午”主效应。下面我们来探索这些显著效应。
1. 点击“拟合混合模型”红色小三角菜单,然后选择边缘模型推断 > 刻画器。
通过“边缘模型刻画器”报表(Figure 8.21),您可以查看治疗、月和上午/下午的各种设置对胆固醇水平 (Y) 的效应。
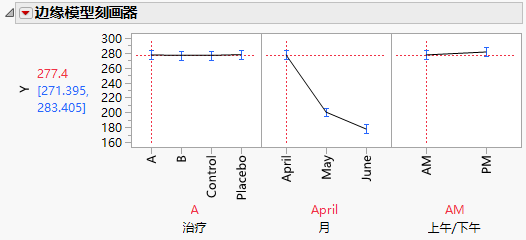
2. 在月图中,将红色的垂直虚线从“April”拖到“May”,然后拖到“June”。
请注意预测的 Y 上午测量值在三个月内是下降的,从四月的均值 277.4 降到六月的均值 177.7。
3. 在治疗图中,将红色的垂直虚线从 A 拖到 B。
通过在月图中将线从“April”拖到“June”,您看到对于治疗 B,预测的 Y 上午测量值从四月的 276.8 降到六月的 191.2。
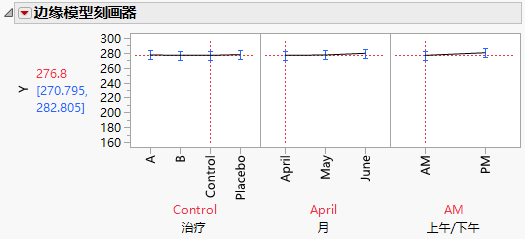
4. 在治疗图中,将垂直虚线拖到“Control”,再拖到“Placebo”。
请注意,当您将治疗设置为“Control”或“Placebo”时,发现胆固醇水平在这三个月中没有什么变化(Figure 8.22)。
接着,您探索上午/下午的效应。
5. 通过拖动红色的垂直线,将治疗和月设置为其水平的全部 12 个组合。
对于这 12 个组合,预测的胆固醇水平在下午都高于早上的值,这演示了主效应。
请注意,治疗 A 在五月得到的胆固醇读数比治疗 B 低。若该效应是显著的,这可能指示治疗 A 的效果比 B 快。下一节(比较六月的所有治疗)说明如何评估治疗。
图 8.21 治疗 A 的边缘刻画器图
图 8.22 Control 的边缘刻画器图
比较六月的所有治疗
在四月、五月和六月这三个月进行研究。您想知道哪些治疗在六月下午的测量值存在差异。
1. 点击“拟合混合模型”红色小三角并选择多重比较。
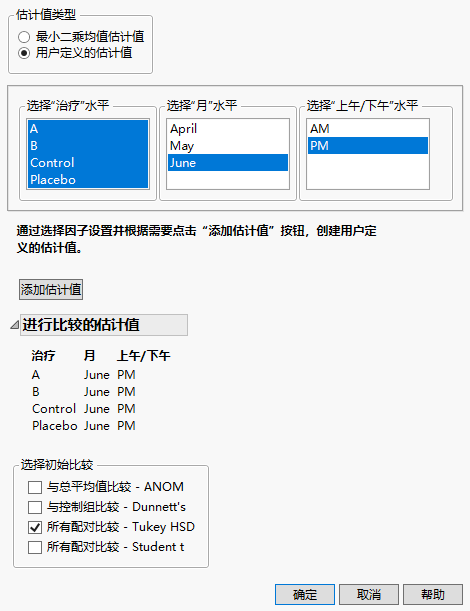
2. 在“估计值类型”下,选择用户定义的估计值。
3. 从“选择‘治疗’水平”面板,选择所有四种治疗类型。
4. 从“选择‘月’水平”面板,选择“June”。
5. 从“选择‘上午/下午’水平”面板,选择“PM”。
6. 点击添加估计值。
7. 从“选择初始比较”列表,选择所有配对比较 - Tukey HSD。
图 8.23 完成的“多重比较”窗口
8. 点击确定。
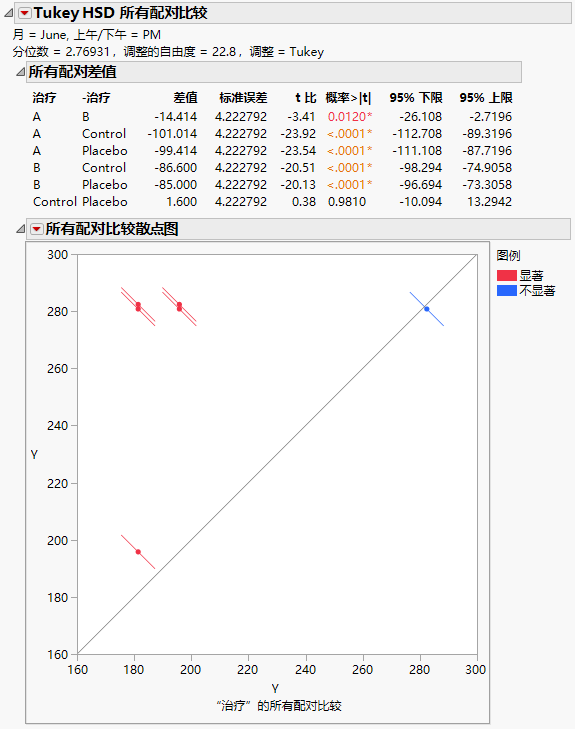
图 8.24 六月下午所有治疗的“Tukey HSD 所有配对比较”报表
“Tukey HSD 所有配对比较”报表显示“所有配对差值”报表和“所有配对比较散点图”。在六月下午的测量值中,除了“Control”和“Placebo”之外的所有治疗均存在显著差异。
考虑治疗 A 和 B 之间的差值。均值差值为 -14.414,置信区间范围为 -26.108 到 -2.7196。您得到结论:治疗 A 导致的胆固醇测量值的减少超过治疗 B 导致的减少大约在 2.7 点到 26.1 点之间。治疗 A 和 B 与“Control”以及“Placebo”相比都是高度有效的。
 AR(1) 模型的回归模型示例
AR(1) 模型的回归模型示例
使用“月”和“上午/下午”分类效应,您比较了胆固醇数据的四个协方差结构。(请注意分类效应对于“非结构化”拟合是必需的。)您已决定使用“AR(1)”协方差结构。
现在假定您要根据连续效应“天数”而非分类效应对治疗效应建模。然后您可以预测治疗期间内的任意时间的胆固醇水平。
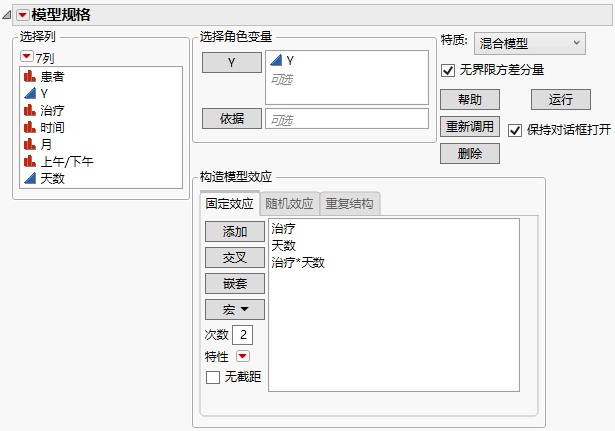
1. 在执行协方差结构:AR(1)中的step 1到step 4后,返回到“拟合模型”启动窗口。
2. 在“固定效应”选项卡上,选择现有的固定效应,然后点击删除。
3. 选择治疗和天数,然后选择宏 > 完全析因。
图 8.25 显示“固定效应”选项卡的“拟合模型”启动窗口
4. 点击“模型规格”红色小三角并取消选择中心多项式。
注意:在默认设置中,交互作用项中使用的连续效应被中心化。通过禁用“中心多项式”选项,交互作用项中使用的连续效应不会被中心化。
5. 点击运行。
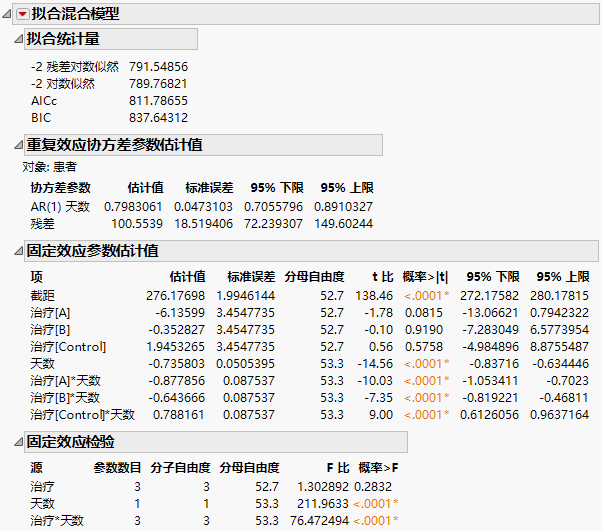
“拟合混合模型”报表显示在Figure 8.26 中。您看到“治疗”和“天数”的交互作用是高度显著的,这指示药物的回归存在差异。
注意:要预测不同天数后药物的效果,请使用刻画器。请参见《刻画器指南》中的刻画器。
图 8.26 “拟合混合模型”报表 - 具有连续固定效应的“AR(1)”协方差结构